核酸・タンパク質複合体
「ヒト生細胞中の核酸はもっとアクティブ!― 試験管内とは違うリアルな動きとは? ―」
■背景
私たちの体の細胞の中には、DNAやRNA、タンパク質などの生体分子が 1リットルあたり400g も詰まっており、とても混み合っています。そんな環境の中で、核酸(DNAやRNA)はピコ秒からミリ秒という速さで構造を変えることで、遺伝情報の読み取りや遺伝子のスイッチのオン・オフをコントロールしていると考えられています。これまで、こうした分子の働きは 試験管の中(希薄な水溶液)で研究されてきました。しかし、ヒト生細胞の中でも同じように動いているのでしょうか? もし違うなら、私たちがこれまでに学んできた「核酸の動き」に関する理解を見直す必要があるかもしれません。
この疑問を解明するために、私たちは 核磁気共鳴(NMR)分光法という手法を活用しました。NMRは、タンパク質や核酸の立体構造や動きを詳しく調べることができる技術です。このNMRを生きた細胞の中に応用したin-cell NMR(インセルNMR)を使い、ヒト生細胞内での核酸のリアルな動きを観察しました。
DNAやRNAは、遺伝情報を持つだけでなく、ときには特殊な立体構造を作りながら遺伝子の働きを調整しています。たとえば、DNAの二重らせんは「ワトソン・クリック型塩基対」と呼ばれる塩基間の水素結合によって作られますが、それ以外にも「フーグスティン型塩基対」などがあり、それによって三重らせんや四重鎖といった構造も形成されます。これらの構造は、遺伝子のスイッチを切り替える重要な役割を果たしています。核酸結合タンパク質が関与するケースも多くあります。私たちはこれまで、二重らせん、三重らせん、核酸―ペプチド複合体が生細胞内でも形成されることをインセルNMRで示してきました(文献1-5)。今回、ヒト生細胞の中で核酸がどのように動いているのかをインセルNMRで調べることにしました。
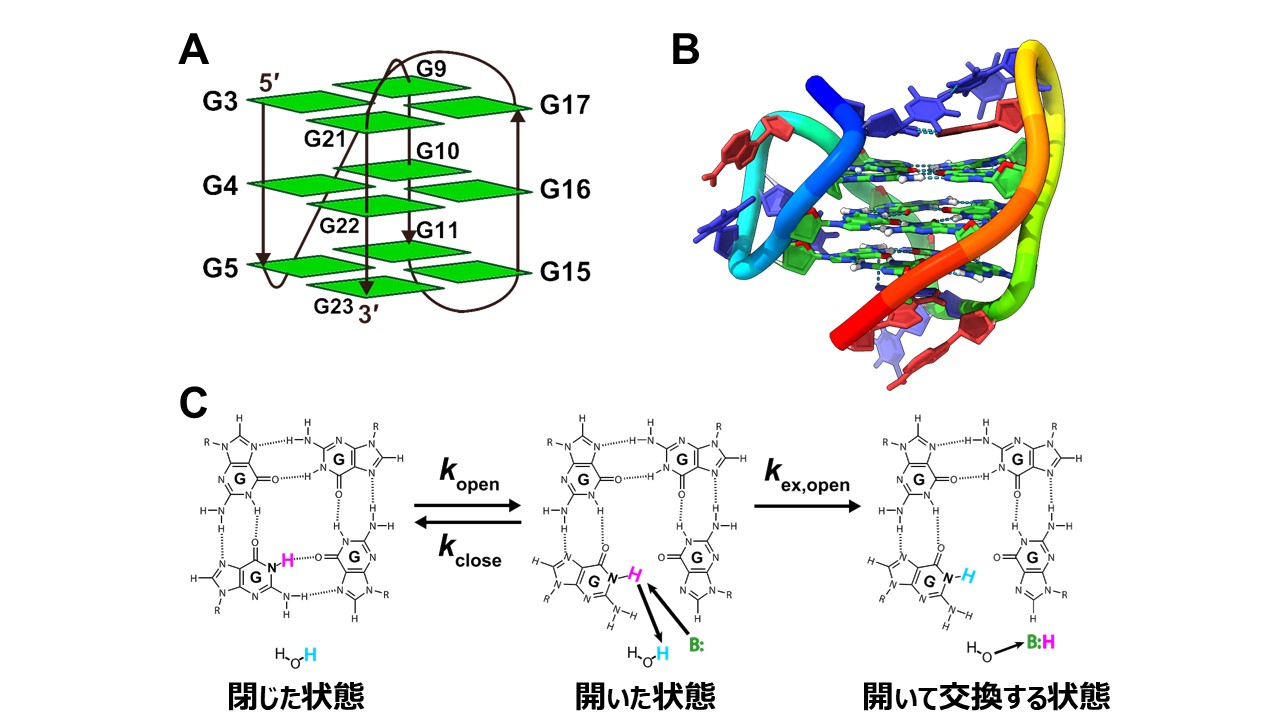
図1 (A, B) ヒト染色体末端に見られるテロメアのモデル配列(5′-TTGGG(TTAGGG)3A-3′)が形成する四重鎖構造。模式図(A)と試験管内実験で決定されたNMR構造(PDB ID: 2GKU) (B)。4つのグアニン塩基(C:緑;H:白;N:青;O:赤)がフーグスティン型塩基対を形成し、1枚のGカルテットを作っている(テロメアモデル配列には3枚ある)。リン酸骨格はイモムシ表示(5′末端(青)から3′末端(赤)まで虹色表現)。アデニン塩基(赤)、チミン塩基(青)、水素結合(水色点線)。(C) Gカルテットの「閉じた状態」では、イミノプロトン(H:桃)が水素結合を形成している。「開いた状態」では水素結合が切れており、塩基触媒(B::緑)がイミノプロトンに近づくことができる。「開いて交換する状態」では、イミノプロトンは塩基触媒に引き抜かれ、水プロトン(H:水色)と化学交換する。kopen, kclose:グアニン塩基対の開閉速度定数。kex,open:開いた塩基対からイミノプロトンが水プロトンと交換する速度定数。
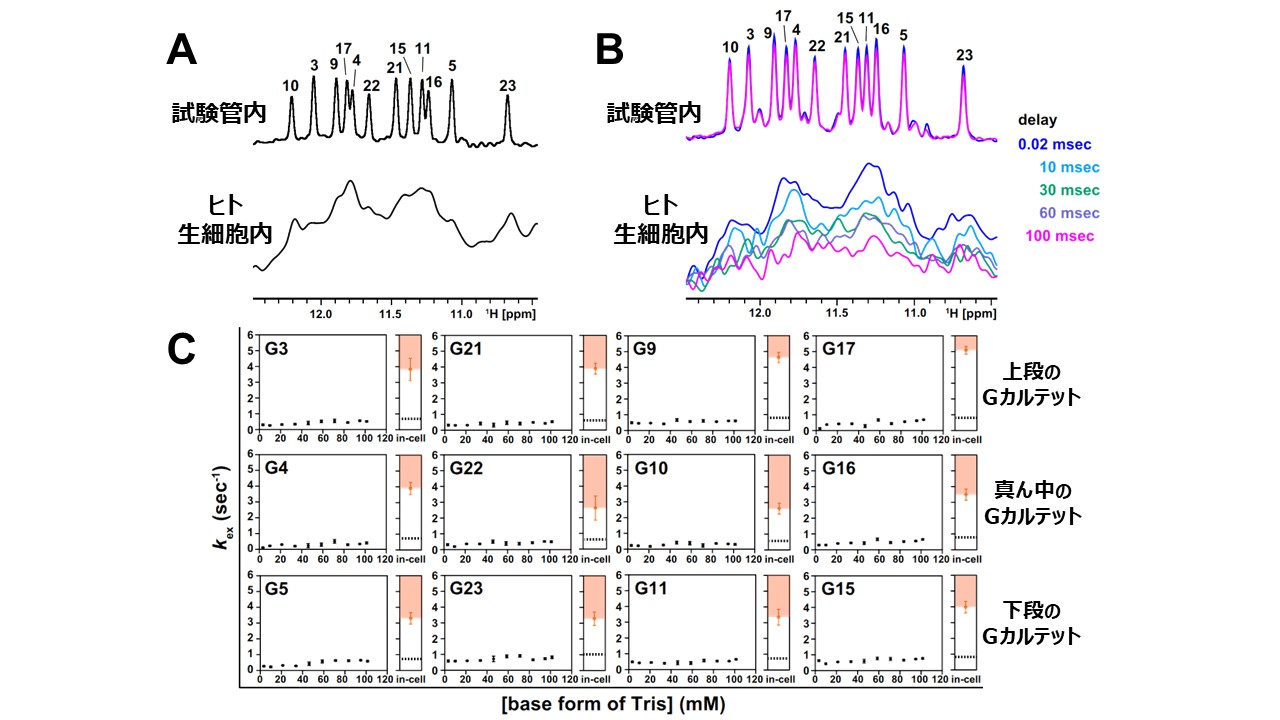
図2 (A上) 試験管内で測定されたテロメアモデル配列のNMRスペクトル。グアニン塩基のイミノプロトンのシグナルが観測された(図中の番号は図1Aの残基番号に対応する)。シグナルが観測されるのは、イミノプロトンが水素結合を形成しているから。(A下) ヒト生細胞内に導入されたテロメアモデル配列のNMRスペクトル。A上で観測されたシグナルに対応する位置にシグナルが観測された。つまり、ヒト生細胞内でも四重鎖構造が形成されている。(B) イミノプロトン―水プロトンの化学交換速度定数(kex)を算出するためのNMRスペクトル。待ち時間の間に化学交換が起こるとシグナル強度が減弱する。シグナル強度の減弱の度合いは試験管内よりもヒト生細胞内の方が明らかに大きいことがわかった。(C) 試験管内において、塩基触媒(base form of Tris)の濃度が高い条件下で得られたkexはkopenに等しい(kex [試験管内] = kopen [試験管内])。一方、ヒト生細胞内の塩基触媒の濃度はわからないが、kex [細胞内] ≥ kopen [細胞内]だと考えられる。したがって、Cのグラフはkopen [細胞内] > kopen [試験管内]であることを示している。つまり、ヒト生細胞内においてテロメアモデル配列はより高い頻度で開いた状態になることがわかった。(文献3)
■研究概要 私たちは、ヒト生細胞内に導入した核酸のNMRシグナルをインセルNMRにより直接観測し、その動きを調べました。その結果、ヒト生細胞内では試験管内よりも核酸の塩基対が頻繁に開くことを見出しました。またこの現象は、細胞内のタンパク質が核酸と非特異的に相互作用することで引き起こされていることもわかりました。図1、図2では、一例としてヒト染色体末端に見られるテロメアのモデル配列(5′-TTGGG(TTAGGG)3A-3′)が形成する四重鎖構造に関する解析を示しました(文献3)。
■科学的・社会的意義 これらの発見は、遺伝子の働きや生命の仕組みを理解するうえで重要な知見となります。さらに、RNAワクチンなどの核酸を利用した医薬品の設計においても、細胞内環境を考慮する必要があることを示唆しています。細胞内での核酸のリアルな動きを正しく理解することで、未来の医療やバイオ技術の発展につながる可能性があります。
■参考文献
1)Eladl, O., et al. (2023). "Complex Formation of an RNA Aptamer with a Part of HIV-1 Tat through Induction of Base Triples in Living Human Cells Proven by In-Cell NMR." Int. J. Mol. Sci., 24(10): 9069.
2)Eladl, O., et al. (2022). " Detection of interaction between an RNA aptamer and its target compound in living human cells using 2D in-cell NMR." Chem. Commun., 59(1): 102-105.
3)Yamaoki, Y., Nagata, T., et al. (2022). "Shedding light on the base-pair opening dynamics of nucleic acids in living human cells." Nature Commun. 13(1): 7143.
4)Sakamoto, T., et al. (2021). "Detection of parallel and antiparallel DNA triplex structures in living human cells using in-cell NMR." Chem. Commun. 57: 6364-6367.
5)Yamaoki, Y., et al. (2018). " The first successful observation of in-cell NMR signals of DNA and RNA in living human cells." Phys. Chem. Chem. Phys. 20(5): 2982-2985.
■良く使用する材料・機器
1) 核磁気共鳴装置 AVANCE-III HD 600、AVANCE-III HD 800、(ブルカー・バイオスピン株式会社)
2) タンパク質精製装置AKTA Go(cytiva)
3) 研究用システム顕微鏡 BX51(オリンパス株式会社)
4) 安定同位体(2H,13C,15N)標識試薬(大陽日酸株式会社)
5) 実験試薬(和光純薬株式会社、ナカライテスク株式会社)
 2025年分野別専門委員
2025年分野別専門委員
京都大学エネルギー理工学研究所
永田崇 (ながたたかし)
https://www.iae.kyoto-u.ac.jp/bio/
「ゲノムサイズのDNAの示す特異な性質:折り畳み転移」
■背景
ヒトの細胞核には48本のDNAが存在し、各々のDNA鎖は数cm、その全てをつなぎ合わせると全長は2mに及ぶ。塩基対をモノマーの単位とすると、60億(6・109)のモノマーから成り立っています(体細胞は2倍体なので、一つの細胞内でDNAが保持している情報は30億塩基対)。わずか数µmのサイズの空間(細胞核)にこのような長い高分子が存在し、生命の基本的情報を担っています。バクテリアでも、DNAは数mm程度と極めて長い。一方、これまでのDNA研究ではゲノムDNAを,全長が1mm以下のサイズにまで断片化してその構造や活性を調べることが専ら行われてきており、長いDNA分子が持っている性質については未だ不明な点が多く残されています。
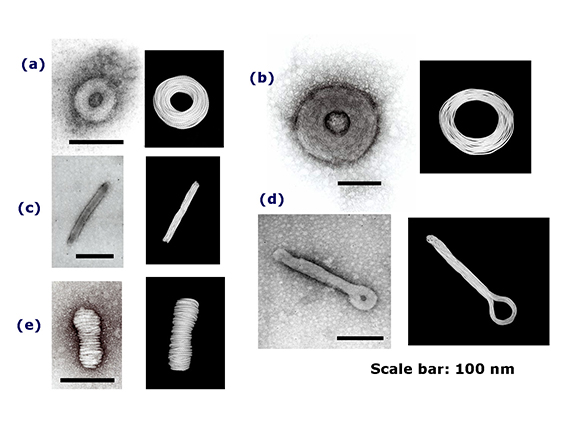
図1: 長鎖DNA(16万5千塩基対、全長57mm)の折り畳み構造の多様性。 各々の図の左は透過電子顕微鏡像(スケール参照)、右は紐で作った「実空間モデル」。 a) ドーナツ、b)巨大ドーナツ、c)丸棒、d)円と棒の組み合わせ、e)糸巻。[ 図は以下の論文参照。a,b)Y.Yoshikawa, et al., Langmuir, 15, 4085(1999); c,d) K. Yoshikawa, et al., Chem.Phys.Lett., 354, 354(2002); e) Y. Yoshikawa, et al., FEBS Lett., 396, 71(1996). ]
■研究概要
図1に示したように、同一の長いDNA分子(16万5千塩基対、全長57mm)1分子から、多様な凝縮構造を創りだすことが可能であることが明らかになりつつあります。これらの構造を大別しますと、液体、結晶、ガラス(アモルファスと言っても良い)に相当する構造とっていることが分かります。

図2 定常期(stationary phase)にある大腸菌からゲノムDNAの光ピンセットで引き延ばしている様子: 蛍光顕微鏡像(▽は光トラップしている場所)と蛍光強度の疑似3次元像。全体の観測時間は13秒。(Shindo, et al., J. Biotech., 133, 172(2008)).
図2には、顕微鏡のステージ上で、大腸菌を溶菌させ、そこからでてきたゲノムDNAをレーザで引き延ばした実験例です。 ここでは、細胞分裂の停止した静止期(stationary phase)の大腸菌での実験例を示しています。実際の実験では、溶出したゲノムDNAの一部にレーザの焦点を固定しておき、試料溶液を載せたスライドガラスを矢印の方向に動かすことにより引き延ばしています。下段に行くにつれて凝縮したDNAが次第に引き延ばされている様子がわかると思います。明るい光の塊(DNAが硬く凝縮している状態)が分裂し、その塊の間には線状に引き延ばされたDNA鎖が観測されている。すなわち静止期において、大腸菌に存在するゲノムDNAには、硬く凝縮した部分と、引き延ばし可能な緩く凝縮した部分が存在していたことを、意味しています。一方、増殖期の大腸菌で同様の引き伸ばし実験をしてみると、ゲノムDNA全体が容易に解け、硬く凝縮した部位は見られません。静止期の大腸菌は、生存に必要な遺伝子群(house-keeping genes)のみを発現しており、その他の多くの遺伝子は完全なoff状態になっていると考えられますが、図2に示したような実験結果は、遺伝子群のon/off制御に、長鎖のゲノムDNAの部分相分離が直接関わっていることを示唆するものとなっています。
■科学的・社会的意義 ヒトの個々の細胞には、2万個を越える遺伝子があり、全長2m程度のDNA分子での塩基の配列としてそれらの情報がきざまれています。私たちの体は単一の受精卵から細胞分裂を繰り返し、細胞が分化するなかで、創りあげられてきていますが、その根本的なメカニズムは不明のままです。iPS細胞やES細胞を用いた再生医療が発展するためにも、細胞の多数の遺伝子群のon/offの制御機構を明らかにすることは基本的で不可欠の研究課題となっています。
■参考文献
1)"生物学は「発見の時代」から「生命の本質に迫る時代」へ"、吉川 研一著, Newton, 7 (2013).
2)"単一高分子の自己秩序形成", 吉川 研一著, 高分子学会, 63, 838 (2014).
■良く使用する材料・機器
1) 蛍光顕微鏡システム (株式会社ニコンインステック、オリンパス株式会社、カールツァイス株式会社)
2) 高感度顕微鏡カメラ (浜松ホトニクス株式会社)
 H27年度分野別専門委員
H27年度分野別専門委員
同志社大学 吉川研一 (ヨシカワ ケンイチ)
https://dmpl.doshisha.ac.jp/
