構造生命情報科学
「コンピュータによるタンパク質のシミュレーション」
■背景 「構造生命情報科学」は、タンパク質やDNA、糖といった生体分子の立体構造をコンピュータで解析する分野で、生物学や情報学だけでなく化学や物理学も必要とする複合的な研究領域に属します。ここでは、タンパク質の運動性を分子動力学シミュレーションにより解析する物理化学的な研究についての研究概要(使用機材、計算結果例)や意義を説明します。

図1 GPGPU搭載の計算サーバー
A: 表側。ハードディスク(HDD)や無停電電源装置(UPS)がある。B: 裏側。1台のサーバーにGPGPUが8機搭載されている。
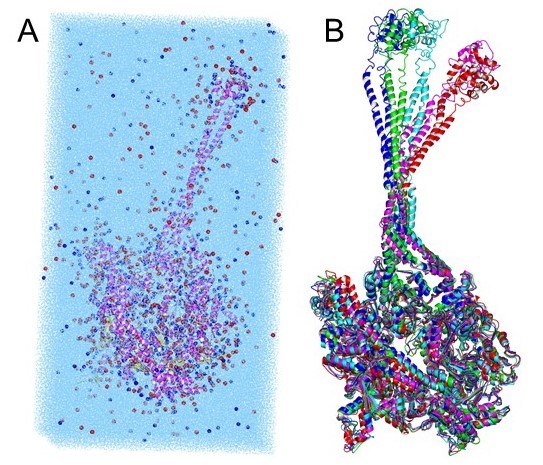
図2 ダイニンモータードメインの分子動力学シミュレーション
A: 約百万原子から成る計算系。ダイニン(リボン)、ATP、水(水色)、イオン(球)から構成される。B: ダイニンの200 nsの分子動力学シミュレーションから得られた構造。赤40 ns、緑80ns、青120ns、紫160ns、水色200ns。水分子やイオンは図から除いた。
■研究概要 タンパク質の計算には、量子化学計算や分子力学計算、分子動力学シミュレーション、ドッキング計算などがあり、これらは創薬研究に応用されています。また創薬を志す学生のための教科書も出版されています(文献1)。著者はタンパク質の分子動力学シミュレーションに関する研究を行っています。具体的には、図1のコンピュータを使ったタンパク質構造の時間変化のシミュレーションです。このコンピュータはPC (通常のコンピュータ)にGPGPUと呼ばれる汎用のグラフィックボードを搭載した構成で、計算時間がかかる静電相互作用部分の計算をGPGPUで超並列化することで高速計算を実現しています。例えば、約百万原子から成る巨大なダイニンの系においても数か月ぐらいの計算で分子運動が解析できます。この分子動力学シミュレーションから、ダイニンのストーク(棒状の構造をした部分)が大きく動くことがわかります(図2)。
■科学的・社会的意義 タンパク質の運動を実験で直接観測することは困難であるため、タンパク質の運動を原子レベルでシミュレーションすることには大きな科学的意義があります。シミュレーションやデータベースをうまく使えば、薬剤を低コストでデザインすることができ、社会的にも期待されています。
■参考文献
1)神谷成敏、肥後順一、福西快文、中村春木 「タンパク質計算科学 基礎と創薬への応用」 共立出版 (2009).
2)N. Kamiya, T. Mashimo, Y. Takano, T. Kon, G. Kurisu, H. Nakamura "Elastic properties of dynein motor domain obtained from all-atom molecular dynamics simulations" Protein Eng. Des. Sel. 29, 317-325 (2016).
■良く使用する材料・機器
1) GPGPU計算サーバー (HP、NEC)
2) Linux OSのパソコン (パソコン工房)
3) ハードディスク (IO DATA)
H28年度分野別専門委員
兵庫県立大学大学院・シミュレーション学研究科
神谷 成敏 (かみや なるとし)
https://sites.google.com/site/cvofnkamiya/
