分子進化・タンパク質進化
「計算による祖先(絶滅)遺伝子・タンパク質の再現」
■背景
生物は37億年の歳月を経て進化してきました。これは、それらの生物を形作る遺伝子・タンパク質の進化でもあります。しかし、太古に存在した遺伝子はすでに存在しません。それらの「絶滅遺伝子・タンパク質」の姿形や機能を知る方法はないのでしょうか?最近では、情報生物学(バイオインフォマティクス)と分子生物学の進歩に基づいた、計算により再現する方法があります。
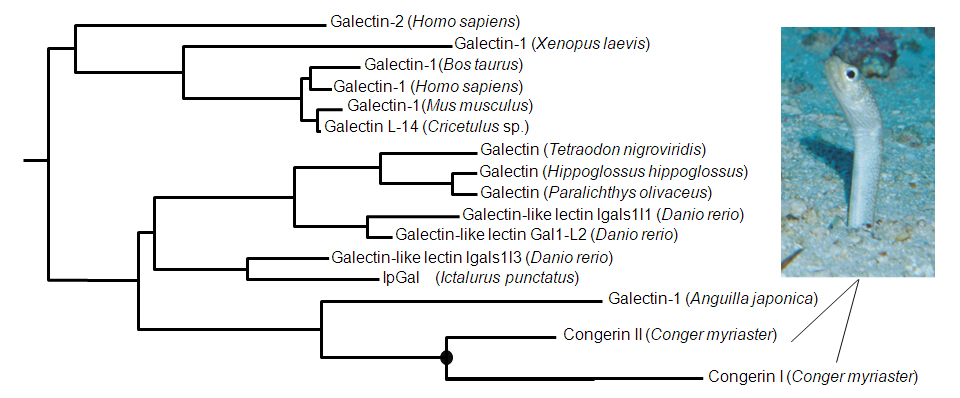
図1 分子系統樹。遺伝子・タンパク質の配列の差 = 進化(分岐後の時間)距離として、各種生物の対応する(相同な)遺伝子・タンパク質の分岐を推定し、樹状に表現したもの。この系統樹の枝の結節点(ノード)には、そこから派生した分子の祖先となる遺伝子・タンパク質が過去に存在しています。。一例として図中黒丸がCongerin IとIIの祖先です。ベイズ推定という統計学の手法を用いて、現存する分子の配列からこれらノードに位置する祖先配列を推定できます。

図2 分子系統樹による祖先型遺伝子推定の模式図
現在の塩基配列を観測データとして、祖先ノードの塩基配列を推定します。図の例では、祖先1と2はG(グアニン)で現在の塩基を説明できます。ただし、祖先3はG(グアニン)でもC(シトシン)でも1回の塩基置換(矢印)で説明できるので、これらの塩基はほぼ同等の尤度(もっともらしさ)を持ちます。実際は示されていないA(アデニン)やT(チミン)の塩基も含めて尤度を計算します。
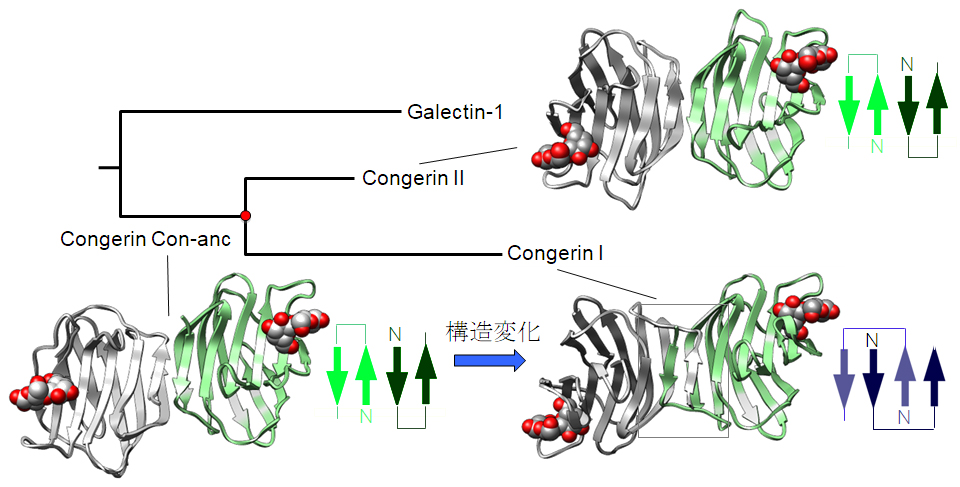
図3 X線結晶解析を用いて解明した、Congerin I、Congerin II、祖先型Congerin (Con-anc)の立体構造(それぞれのタンパク質でサブユニットを白と緑で表示しています)。Congerin Iの中央付近に長方形で示した領域のβシート構造が、矢印の模式図で示したようにCon-ancからCongerin Iの間で変化しています。一方、Congerin IIは祖先型の構造を受け継いでいます。
■研究概要
魚類の生体防御に関与するCongerin(コンジェリン)というタンパク質は、アナゴ(Conger myriaster)などではCongerin IとCongerin IIという、2つの異なる遺伝子・タンパク質に分化していて、それぞれ機能や構造が少し異なっています(図1)。特にCongerin IではIIつのサブユニット(生体内では同じCongerinが2個組み合わされて働いています。このとき、それぞれをサブユニットと呼びます)の間でペプチドが絡み合わされて、サブユニットがはずれにくい様に構造が強化されています。祖先型Congerin (Con-anc)のアミノ酸配列を系統樹に基づいてベイズ推定で予測し(図2)、実際にタンパク質を作成し、構造解析したところ、祖先型CongerinはCongerin Iのようなペプチドの絡み合わせ構造を持たず、構造的にも機能的にもCongerin IIに近いことがわかりました(図3)。これは、Congerin Iは遺伝子重複によりCongerin IIと分岐することで、大きく構造変化した強化型に進化できたことを示しています。
■科学的・社会的意義
天然のタンパク質を、より産業的価値を高めるように人工的に改変する技術を、タンパク工学と呼びます。生物の進化は、天然の非常に成功したタンパク工学と考えられます。祖先型の遺伝子・タンパク質を再現して、構造や機能を解析することで、すでに失われた優れた性質を持った遺伝子を発見したり、その過程でタンパク工学の技術を洗練させることができます。
■参考文献
1)Konno, A., Kitagawa, A., Watanabe, M., Ogawa, T., Shirai, T. (2011). “Tracing protein evolution with ancestral structures of fish galectin.” Structure 19: 711-722.
■良く使用する材料・機器
1) ワークステーション(高速計算機)
2) X線回折装置(主にSpring8やPhoton Factory/ARなどの放射光)
 H29年度分野別専門委員
H29年度分野別専門委員
長浜バイオ大学・バイオサイエンス学部
白井 剛 (しらいつよし)
https://www.nagahama-i-bio.ac.jp/research/
「タンパク質進化における多様性」
■背景
タンパク質は20種類のアミノ酸がいろいろな順序で結合した1本の糸(ポリマー)です。この組み合わせはアミノ酸100個という短いタンパク質でも10130種類という宇宙全体の原子の数より大きな天文学的数字になります。
例えば、人間には約10万種類のタンパク質があると言われています。
そのタンパク質ひとつひとつはその機能に関して最適なたった一つの配列といえるのでしょうか。それとも複数の解をもちえるのでしょうか?
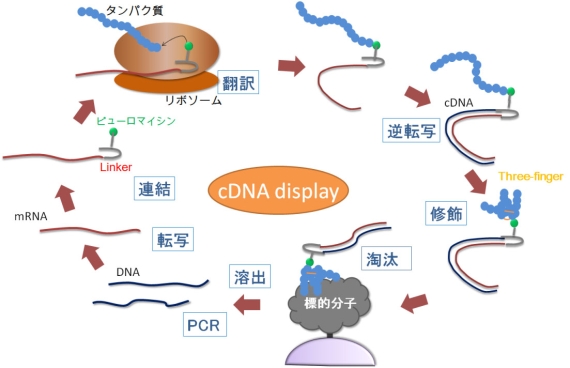
図1 進化分子工学のcDNA display法による試験管内淘汰
ランダムなタンパク質を無細胞翻訳中のリボソームで合成し、mRNAをcDNAに逆転写し、cDNAとそれにコードされたタンパク質を連結体(cDNA display分子)のライブラリを標的分子と接触させ、結合能のあるものを淘汰する。
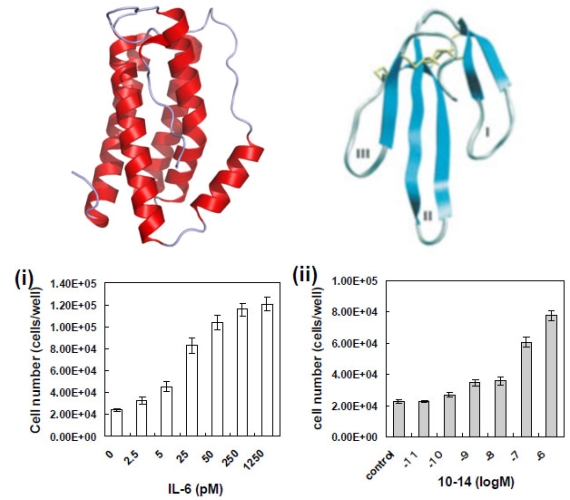
図2 IL-6レセプターにアゴニストとして働くIL-6とThree-finger scaffold
IL-6はαへリックスという2次構造からできている(左)。一方、Three-finger scaffoldはβシートという2次構造からできている(右)。しかし、Three-finger scaffoldの指先部分に変異を入れたライブラリを作製し、図1の方法で淘汰したところ下の細胞増殖アッセイにあるようにIL-6と同じように細胞を増殖させることがわかった。(参考文献2参照)
■研究概要
私たちは1012種類のタンパク質から特定の機能のみを淘汰する進化分子工学の技術を用いて、抗体医薬の標的ともなっているインターロイキン6レセプター(IL-6R)に対し、本来結合しない蛇毒由来タンパク質(Three-finger Toxin)の一部をランダムに変異させたライブラリを用いて特異的に結合するものを複数取得しました。興味深いことにこの中には結合するだけでなく、インターロイキン6(IL-6)と同じように作用するものが見出されました。
■科学的・社会的意義
抗体は分子標的医薬として大変有用なバイオ医薬品として注目されています。しかし、高額な製造コスト、診断チップに搭載する際の安定性などの問題があります。今回用いたThree-finger Toxinは大腸菌で安価に合成でき、抗体に比べ格段に安定性が高いという特徴があります。その指先部分をランダム配列にすることで抗体のように様々な分子を認識することが可能な次世代抗体の一種です。まさに毒を薬に変えるバイオ医薬といえます。従来の抗体の課題を克服し、より広く医療や産業応用に役立つことが期待されています。
■参考文献
1) J. Yamaguchi, M. Naimuddin, M. Biyani, T. Sasaki, M. Machida, T. Kubo, T. Funatsu, Y. Husimi, N. Nemoto, “cDNA display: a novel screening method for functional disulfide-rich peptides by solid-phase synthesis and stabilization of mRNA-protein fusions” Nucleic Acids Res, Vol.37, e108 (2009)
2) M. Naimuddin, S. Kobayashi, C. Tsutsui, M. Machida, N. Nemoto, T. Sakai, T.Kubo, “Directed evolution of a three-finger neurotoxin using cDNA display
yields antagonists as well as agonists of interleukin-6 receptor signaling” Mol.
Brain, Vol.4, 2 (2011)
■良く使用する材料・機器
1)蛍光相関分光法(FCS)装置(和光純薬株式会社)
2)実験試薬 (和光純薬株式会社)
 H24年度分野別専門委員
H24年度分野別専門委員
埼玉大学・工学部機能材料工学科
根本直人 (ねもとなおと)
https://www.fms.saitama-u.ac.jp/lab/nemoto/publication.html










