モチーフ・モジュール・ドメイン
「たんぱく質の形にひそむ法則を探し出す」
■背景
いきものは数多くのたんぱく質が集まってできており、私たち人間では約10万種類ものたんぱく質が働いていると言われています。それぞれのたんぱく質はひとつひとつ違った形をもっており、その形に応じた役割をもっています。とりわけ、多くのたんぱく質には表面に凹凸があり、くぼんだ場所にはちょうどぴったりはまる物質(「リガンド」と呼びます)が存在することがあります。かつてエミル・フィッシャーはこれを「鍵と鍵穴」の関係になぞらえました。ヒトが持つたんぱく質の「鍵穴」にぴったりはまる「鍵」を作り出すことで、たんぱく質を操ることができます。これがまさに薬を創るということになります。しかしこの「ぴったりはまる」ということがそう簡単ではありません。鍵穴の形が非常に複雑かつ多様で、どのようなものが鍵となるのか、見ただけでは分からないからです。
そこで、鍵穴を鍵穴たらしめる特徴を、その普遍的な法則をつまびらかにすることが重要となります。たんぱく質の役割に重要な、多くのたんぱく質に共通してみられる特徴的な形のことを、「モチーフ」と呼びます(厳密な定義は難しいのですが、ここではそういうことにしておきます)。
図1. 鍵と鍵穴の関係となるリガンドとたんぱく質。Aは全体像で、特に鍵穴周辺を拡大したものがBです。水色で描かれたリガンドの周辺に、鍵穴の部品であるアミノ酸が並んでいます。この鍵と鍵穴に見られるパターンを描いたものがC、D、Eです。(図は参考文献1に基づいて改変したもの)
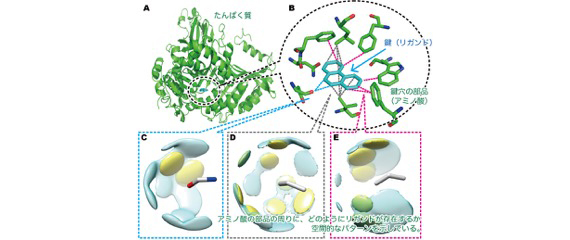
■研究概要 多くのたんぱく質の鍵穴に共通の形である「モチーフ」を探し出し、その普遍的な法則を明らかにすることが研究の目的となります。そのためには様々なたんぱく質の形を調べ、共通性を見つけることが必要です。これまでに世界中の研究者によって様々なたんぱく質の形が調べられており、それは「たんぱく質構造データバンク」(Protein Data Bank; PDB)と呼ばれるデータベースに集められています。私の研究ではこの膨大なたんぱく質構造データを、計算機を使って網羅的に解析し、共通のパターンを見つけ出し、鍵と鍵穴の特徴を調べています。ここでは大規模データを解析するためのプログラムやアルゴリズム開発などの情報科学技術、パターン発見や相関解析などの数理統計技術、物理シミュレーションなどの計算物理技術などを利用しています。
■科学的・社会的意義 ひとつの新しい薬を創るには数百億円の予算が必要と言われています。たんぱく質の形にひそむモチーフを明らかにし、たんぱく質の鍵穴の仕組みを解明できれば、もっと効率的に薬を作れるようになるかもしれません。未解決の問題はまだまだ山積みですが、研究を続けていくことでその成果を社会へ還元していけると考えています。
■参考文献
1) Kasahara K, Kinoshita K, (2016) “Landscape of Protein–Small ligand Binding Modes” Protein Science 25(9):1659-71
2) Kasahara K, Kinoshita K, (2014) “GIANT: pattern analysis of molecular interactions in 3D structures of protein–small ligand complexes.” BMC Bioinformatics 15:12
3) Kasahara K, Shirota M, Kinoshita K (2013) “Comprehensive classification and diversity assessment of atomic contacts in protein-small ligand interactions.” J Chem Inf Model 53:241–248
■良く使用する材料・機器
1) 大型計算機(株式会社日本HP、デル株式会社など)
2) myPresto(一般社団法人バイオ産業情報化コンソーシアム)
 H28年度分野別専門委員
H28年度分野別専門委員
立命館大学・生命科学部
笠原浩太 (笠原浩太)
https://ktkshr.net/
「ドメインを単位としてタンパク質の機能や構造を理解する」
■背景
タンパク質は、たくさんのアミノ酸がヒモの様につながることで作られる生体分子です。多くのタンパク質は、固有の形(立体構造)に折りたたまり、それによって特有の機能を持ちます。数百を超えるアミノ酸がつながったタンパク質の立体構造を見ると、タンパク質全体が毛糸玉のような一つのかたまりに折りたたまれることはあまりなく、多くの場合、100〜200個程度のアミノ酸が折りたたまった構造が、数珠のようにつながっています(図1)。このような100〜200個程度のアミノ酸が折りたたまった部分構造は「ドメイン」と呼ばれ、複数のドメインから作られるタンパク質のことは「マルチドメインタンパク質」と呼ばれます。
ドメインの中には、マルチドメインタンパク質からその部分だけを切り出してきても安定な立体構造を取ったり、それ単独で特定の機能を担うことができたりする場合があります。そのようなドメインはタンパク質の構造や機能の「単位」と考えることができ、たくさんのアミノ酸からなる複雑なタンパク質も、ドメインという単位に分けることで機能や構造を理解しやすくなります。また、生物が進化する過程で、ドメインの組み合わせを変えることで、新しい機能を持つタンパク質を作り出している例も多く見られます。この意味でドメインは進化の単位とも言うことができます。
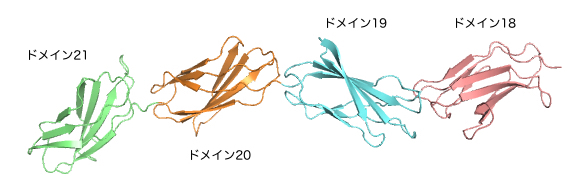
図1 典型的なマルチドメインタンパク質であるヒトのフィブロネクチンの部分構造。ヒトフィブロネクチンは細胞接着などに関わる約2400アミノ酸からなるタンパク質であり、30個のドメインがつながっている。図はそのうちの18〜21番目のドメインを切り出して立体構造を決めたものである(PDB ID: 1FNF、文献1)。図中の4つのドメインはそれぞれ約90アミノ酸から成る。
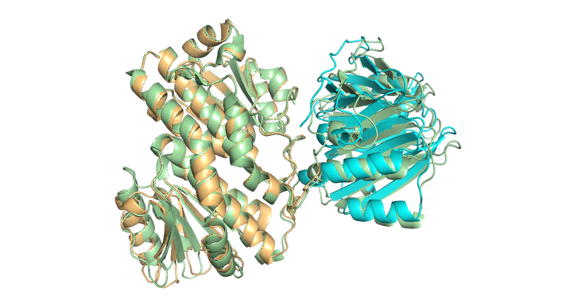
図2 個別に立体構造が決められたドメイン(ベージュとシアン)にもとづいて、グルコサミン6リン酸合成酵素のマルチドメイン構造を予測した結果。緑は2つのドメインがつながった全体構造を実験的に決めたもの。ベージュのドメインがよく重なるように予測した構造を全体構造に重ね合わせた時、シアンのドメインのずれは約2.5Åであり、小さい誤差で全体構造を予測できている。
■研究概要
ヒトゲノムから作られるタンパク質は、平均で約560個のアミノ酸がつながっており、全タンパク質の半分以上がマルチドメインタンパク質と考えられています。これまでは、複雑なマルチドメインタンパク質を理解するために、タンパク質を構成する単位であるドメインごとに機能を解析することが多くなされてきました。しかし、複数のドメインが共同的に働くことで機能が発揮されるマルチドメインタンパク質も多く見られ、そのようなマルチドメインタンパク質の機能を詳細に理解するには、複数のドメインが集合してどのような立体構造を取るかを知ることが重要になります。マルチドメインタンパク質は、実験的な手法の制約から、全体構造を決定するのは容易ではありません。一方で、切り出された個別のドメインの立体構造を決定することは比較的容易になってきており、ドメイン単独の立体構造情報は非常に多く存在しています。
そこで我々は、個別のドメインの構造から、全体構造を予測する方法の研究をしています。これまで、コンピュータを使って、ドメイン間の接触部位にあるアミノ酸の特徴解析や、ドメインどうしをつなぐドメインリンカーの構造の解析を行い、その結果を取り入れた予測アルゴリズムを作成してきました。その結果、2つのドメインからなる構造については既存の方法よりも良い精度で予測することができるようになっています(図2、文献2)。
■科学的・社会的意義 遺伝子の塩基配列決定技術が進歩し、疾病に関連すると考えられている様々な変異が見つかっています。これらの変異がなぜ疾病に結びつくのかを理解するには、タンパク質の立体構造上、どこにその変異が生じているかを知ることが重要です。マルチドメインタンパク質において変異と疾病の関係を理解するには、個々のドメインにおける変異の位置だけでなく、ドメインどうしの相互作用に影響を与える変異かどうかも知る必要があり、それには全体構造の情報が必要となります。実験的に決定することが難しいマルチドメインタンパク質の全体構造を精度良く予測できれば、このような研究にも貢献することができます。
■参考文献
1) Leahy DJ, Aukhil I, Erickson HP. (1996) 2.0 A crystal structure of a four-domain segment of human fibronectin encompassing the RGD loop and synergy region. Cell 84:155-164
2) Hirako S, Shionyu M. (2012) DINE: A novel score function for modeling multidomain protein structures with domain linker and interface restraints. IPSJ Transaction on Bioinformatics 5:18-26.
■良く使用する材料・機器
1) ハイパフォーマンスコンピュータ(日本ヒューレット・パッカード株式会社)
2) PCクラスター(デル株式会社)
3) MOE(CCG、株式会社菱化システム)
4) Discovery Studio(ダッソー・システムズ・バイオビア株式会社、ダイキン工業株式会社・COMTEC )
 H26年度分野別専門委員
H26年度分野別専門委員
長浜バイオ大学・バイオサイエンス学部
塩生真史 (しおにゅうまさふみ)
https://b-lab.nagahama-i-bio.ac.jp/~m_shionyu/index.php/
